
Quando lidamos com dados precisamos, muitas vezes, visualizar de forma gráfica esses dados. Em muitas tarefas é útil, ou até mesmo essencial, que as conclusões das análises sejam mostradas através de gráficos. Mesmo nas fases iniciais de uma análise, na preparação e limpeza de dados, a visualização é importante visualizar para se compreender padrões, tendências e anomalias, tais como pontos fora da curva. Existem no Python inúmeras bibliotecas para visualização de dados e montagem de gráficos. Matplotlib é o módulo básico para uso em conjunto com o pandas.
A biblioteca é grande, com extensas possibilidades e tem sido usada como base para a elaboração de outros módulos gráficos, como o Seaborn. O aprendizado da biblioteca inteira pode demandar um esforço considerável mas o uso básico, suficiente para muitos projetos, não demanda tanto empenho. Além disso o pandas tem uma vinculação natural com a biblioteca, como veremos.
Instalação
Matplotlib é instalado junto com a distribuição do python via Anaconda. Se você não está usando Anaconda é possível encontrar maiores instruções de instalação no site de Matplotlib.
Um pouco de história
Matplotlib começou a ser desenvolvida em 2003 por John D. Hunter, um neurocientista que usava MATLAB e queria aperfeiçoar a visualização de dados obtidos por meio de EEGs (eletroencelografia) em sua pesquisa sobre epilepsia. Hoje uma a comunidade de desenvolvedores colaboram para manter e aperfeiçoar a biblioteca.
Como muitos usuários e desenvolvedores estavam acostumados ao ambiente do MATLAB, onde todas as funções estão disponíveis globalmente sem a necessidade de importações, o módulo pylab foi desenvolvido. Ele existe para trazer funções e classes do NumPy e matplotlib para o namespace global. Isso significa que o comando from pylab import * em uma sessão significa a importação desses módulos e é desnecessária para quem está acostumado com o estilo do python. Como já vimos a importação de muitos módulos, funções e classes pode provocar conflito entre as partes importadas e os métodos built-in do python.
De fato, o uso de ipython --pylab para quem usa o comando de linha, ou %pylab de dentro do Jupyter, simplesmente faz uma chamada interna à from pylab import *. Nesse sentido se recomenda, para quem trabalha com IPython e Jupiter Notebook, que se use a “mágica” %matplotlib.
Por todos esses motivos usaremos a abordagem usual do python:
» import matplotlib.pyplot as plt » import numpy as np » np.random.seed(444) » # para exibição dos gráficos no ambiente do jupyter notebook usamos » %matplotlib » # para exibição incorporada dentro do notebook » %matplotlib inline » # ou, para exibição dentro do notebook, com controles de zoom e arraste » %matplotlib notebook
Numpy será usado para as contruções de arrays e geração de dados aleatórios. A informação de np.random.seed() serve para que os geradores produzam os mesmos números em seções posteriores, para reproducibilidade. A mágica %matplotlib faz com que os gráficos sejam exibidos. Nesse caso uma nova janela é aberta com uma barra de menus com acesso à ampliação, arraste, gravação em vários formatos (como pdf, jpg, png), e parâmetros do gráfico. Já a inserção de %matplotlib inline faz com que os gráficos fiquem embutidos no próprio notebook e sejam gravados com ele. A janela de controle não aparece. Usando %matplotlib notebook temos o gráfico embutido com acesso à controles de zoom, arraste e gravação em arquivo.
Técnica Básica
O matplotlib pode receber como fonte de dados listas, arrays do numpy, Series e dataframes do pandas. Por exemplo, o código seguinte recebe listas gera as figuras 1 e 2 abaixo:
» %matplotlib inline » plt.plot([0,3,0,5,0,7,0]) » # a figura 1 é gerada » plt.plot([0,1,2,3,4,5,6],[0,3,0,5,0,7,0]) » # o mesmo que antes (figura 1 é gerada) » x = np.arange(101)-50 » y = x**2 » plt.plot(x,y) » # a figura 2 é gerada
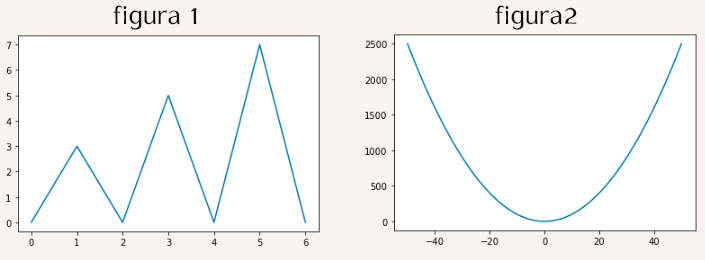
Quando apenas uma lista é fornecida plot usa os índices como coordenada horizontal (abcissa) e os valores da lista como coordenadas verticais (ordenadas). Quando duas listas de mesmo tamanho são fornecidas a primeira é usada para os valores das abcissas, a segunda como ordenadas. No segundo exemplo, que gera a figura 2, foram usadas coordenadas (x, x2) (uma parábola) com x variando no intervalo [-50, 50].
Usando %matplotlib notebook o gráfico é exibido inline mas trazendo controles de ajuste da imagem. Ao se clicar no botão azul (pode ter outra cor na sua instalação) os controles desaparecem e a imagem fica estática.
» %matplotlib notebook
» x = np.arange(40)
» plt.plot(x,np.exp(x/10)) # exibe a figura 3
» # outro exemplo: seno
» # dados a plotar
» x = np.arange(0.0, 2.0, 0.01)
» y = np.sin(2 * np.pi * x)
» fig, ax = plt.subplots()
» ax.plot(x, y)
» ax.set(xlabel='eixo x', ylabel='y = seno(x)', title='Gráfico de seno(2 pi x)')
» ax.grid()
» fig.savefig('seno.jpg')
» plt.show() # exibe a figura 4
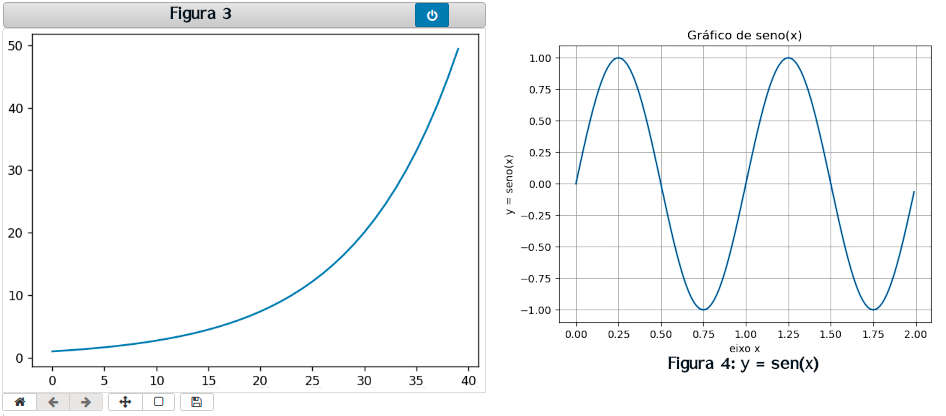
No primeiro caso plotamos simplesmente o gráfico de y = exp(x/10). No segundo exemplo criamos um array no intervalo [0, 2) em passos de .01. A coordenada é y = sen(2πx). Usamos as funções exponencial e seno do numpy para lidar com a operação vetorializada (que pode ser aplicada sobre todo o array). Já veremos com maiores detalhes os métodos de matplotlib.pyplot.
Hierarquia de objetos do Matplotlib
Mesmo em exemplos simples, como os anteriores, Matplotlib usa uma hierarquia de objetos. Por hierarquia se entende que objetos dependem de outros, como em ramos de uma árvore. Repetindo o gráfico da figura 4 temos:
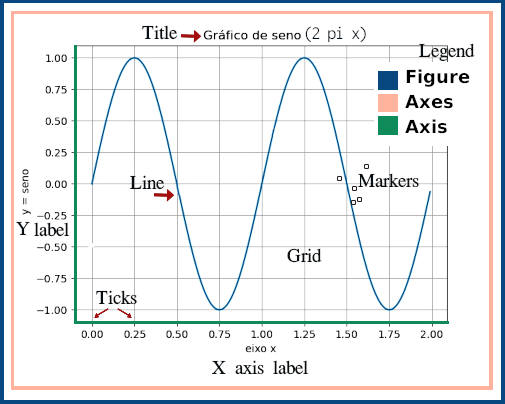
Figure é o objeto básico ou o mais externo de um gráfico. Ele pode conter diversos Axes, que são gráficos ou plotagens individuais†. Axes, por sua vêz, podem conter legendas, marcas gráficas, curvas e caixas de texto. Cada um desses elementos são objetos do python com métodos e propriedades que podem ser manipuladas individualmente.
Vamos verificar em código como esses objetos são criados e manipulados.
» # geramos os dados a imprimir
» x = np.arange(0.0, 2.0, 0.01)
» y = np.sin(2 * np.pi * x)
» # O objeto básico do matplotlib.pyplot (aliás plt) é figure
» fig = plt.figure()
» # De figure derivamos um axes (subplot) e plotamos 3 curvas
» ax = fig.add_subplot()
» ax.plot(x, y)
» ax.plot(x, x)
» ax.plot(x, x**2)
» # definimos os labels dos eixos x e y e o título do gráfico
» ax.set(xlabel='eixo x', ylabel='seno(x), x, x^2',
» title='y = seno, reta e parábola')
» # acrescentamos um quadriculado (grid)
» ax.grid()
» # salvamos a figura no disco
» fig.savefig('seno.jpg')
» # exibimos o resultado
» plt.show()
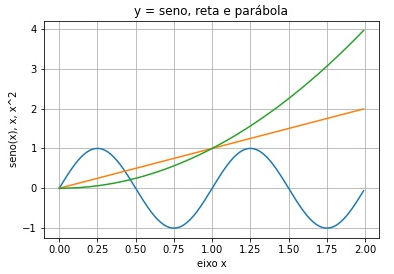
Ao objeto ax acrescentamos 3 plots (y=sen(2πx), y=x, y=x2), os labels dos eixos, o título do gráfico e o quadriculado de fundo. Opcionalmente a figura pode ser salva. A figura só é exibida quando plt.show() é executado.
O método add_subplot admite diversos parâmetros: fig.add_subplot(m,n,r) significa criar um gráfico em m linhas, n colunas, na posição r. Um subplot significa dividir a região destinada ao gráfico em m×n partes onde se pode colocar sub-gráficos.
Por ex., vamos criar uma figura com 4 subplots, plotando uma curva diferente em cada uma delas.
» x = np.arange(0.0, 2.0, 0.01)-1
» fig = plt.figure()
» ax1 = fig.add_subplot(2,2,1) # 2 linhas, 2 colunas: a 1ª figura
» ax2 = fig.add_subplot(2,2,2) # a 2ª figura
» ax3 = fig.add_subplot(2,2,3)
» ax4 = fig.add_subplot(2,2,4)
» ax1.plot(x, np.sin(10*x))
» ax2.plot(x, x)
» ax3.plot(x, x**2)
» ax4.plot(x, x**3)
» fig.savefig('figura5.jpg')
» plt.show()
A mesma figura pode ser obtida fazendo os plots diretamente para os axes:
» fig = plt.figure() » ax1 = fig.add_subplot(2, 2, 1) » plt.plot([-1, 0, 1, 2]) » ax2 = fig.add_subplot(2, 2, 2) » ax3 = fig.add_subplot(2, 2, 3) » plt.plot(np.random.randn(50).cumsum(), 'k--') » ax4 = fig.add_subplot(2, 2, 4) » plt.plot([1.5, 3.5, -2, 1.6])
O método plt.plot(dados) se refere ao eixo ativo que é aquele criado ou usado por último. No caso acima nenhuma figura foi plotada no 2 º retângulo.
O procedimento de criar vários subplots dentro de um mesmo gráfico pode resumido por meio do método
fig, axes = plt.subplots().
subplots() retorna uma tupla onde o 1&orm; elemento é uma Figure, o objeto básico de um plot, e o 2º são os axes que recebem as curvas.
Esses axes podem ser referenciados individualmente pela notação de array. Por ex.: em
fig, axes = plt.subplots(2, 3)
temos axes[0,0] até axes[1,2].
np.random.randn(50).cumsum()
retorna a soma cumulativa dos elementos de um array de 50 elementos “aleatórios”.
pyplot.subplots possui as opções:
| nrows | número de linhas |
| ncols | número de colunas |
| sharex | todos os subplots devem ter os mesmos “ticks” no eixo x |
| sharey | todos os subplots devem ter os mesmos “ticks” no eixo y |
| subplot_kw | dicionário de chaves para criar cada subplot |
| **fig_kw | chaves adicionais, como plt.subplots(2,2,figsize=(8,6)) |
Formatação dos gráficos
Tamanho
O tamanho de uma figura, por default dada em polegadas, é definido pelo parâmetro figsize em plt.figure(figsize=(largura, altura)).
» x = np.arange(.1, 10, 0.01) » largura = 5; altura = 2 » plt.figure(figsize=(largura, altura)) » plt.plot(x, np.log(x)) » plt.show() # gerado o gráfico na figura 8
O mesmo gráfico é gerado usando esse parâmetro no construtor da figure, mas desenhado com linhas pontilhadas, devido ao parâmetro ‘k–‘ em ax.plot(x, np.log(x), 'k--').
» x = np.arange(.1, 10, 0.01) » fig = plt.figure(figsize=(5, 2)) » ax = fig.add_subplot() » ax.plot(x, np.log(x), 'k--') » plt.show() # gerado o gráfico na figura 9
Espaçamento entre subplots, cores e marcadores
O espaçamento entre figuras de um gráfico com subplots, que por default é um espaço não nulo, pode ser ajustado por meio do método Figure.subplots_adjust(). Por conveniência o mesmo método pode ser acessado diretamente pela função:
subplots_adjust((left=None, bottom=None, right=None, top=None, wspace=None, hspace=None).
wspace e hspace indica quanto espaço percentual em relação à largura e altura da figura, respectivamente.
Por exemplo, para juntar os subplots fazemos ambos igual a zero.
» x = np.arange(.1, 10, 0.01) » fig, axes = plt.subplots(2, 2) » axes[0, 0].hist(np.random.randn(1000), bins=100, alpha=.5) » axes[0, 0].hist(np.random.randn(500), bins=50, color='r', alpha=.5) » axes[0, 1].hist(np.random.randn(500), bins=50, color='gold', alpha=1) » axes[1, 0].hist(np.random.randn(500), bins=50, color='#ff0000') » axes[1, 1].plot(x, 3*np.log(x), color='#55aaff') » axes[1, 1].plot(x, x, color='#000000') » plt.subplots_adjust(wspace=0, hspace=0)
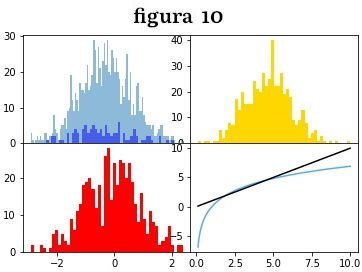
Para usar os mesmos eixos em todos os 4 gráficos usamos os parâmetros sharex, sharey, em
fig, axes = plt.subplots(2, 2), sharex=True, sharey=True).
Em hist(np.random.randn(1000), bins=100) traçamos o histograma de dados aletórios (100 números) separados em 100 bins. No 1º axes traçamos 2 histogramas, o 1º com a cor azul default, o 2º com color=’r’, um atalho para ‘red’ ou vermelho, com transparência de 50%, alpha=.5. Uma lista de cores nomeadas, como color=’gold’ pode ser encontrada no site do matplotlib. Também podemos usar o código de cores html que consiste em 3 números hexadecimais de 0 até 255 (ou 00 até ff em hexadecimal), no sistema rgb (vermelho, verde, azul). Diversos editores de imagens ou de código disponilizam um seletor de cores que retorna a cor nesse sistema. O site HTML-COLOR.CODES também tem um seletor online.
O método axes.plot(), além de aceitar arrays para abcissas e coordenadas, pode receber também o string especificador de cor e tipo de linha. Para imprimir em verde (‘green’) com linha tracejada usamos
ax.plot(x, y, 'g--'),
que é uma forma resumida de passar parâmetros. Isso é o mesmo que:
ax.plot(x, y, linestyle='--', color='g').
Considerando que x e y são arrays de mesmo tamanho, alguns exemplos desses parâmetros são:
» plot(x, y) # plot x, y com linha e estilo default » plot(x, y, 'bo') # plot x, y com marcadores azuis circulares » plot(x, y, 'rv') # plot x, y com marcadores vermelhos, triângulo para baixo » plot(y) # plot y usando seus índices como coordenadas-x » plot(y, 'r+') # idem, usando cruzes vermelhas
No Jupyter Notebook use plt.plot? para ver uma lista completa dos parâmetros desse método.
Ao desenhar um gráfico pode ser interessante marcar os pontos sobre as curvas contínuas. Isso é feito com markers ou marcadores.
» from numpy.random import randn » plt.plot(randn(30).cumsum(), color='green', linestyle='dashed', marker='o')
Uma forma abreviada para o mesmo comando é: plt.plot(randn(30).cumsum(), 'go--'), onde os parâmetros são passados em uma única string, com g para green (verde), o para o marcador circular e -- para o estilo de linha tracejado.
Por default os pontos de um plot são ligados por linhas. Para outro estilo usamos drawstyle:
» data = np.random.randn(20).cumsum() » plt.plot(data, 'b--', label='default', marker='v') # linha azul » plt.plot(data, 'r-', drawstyle='steps-post', label='passos') # linha vermelha » plt.legend(loc='best')
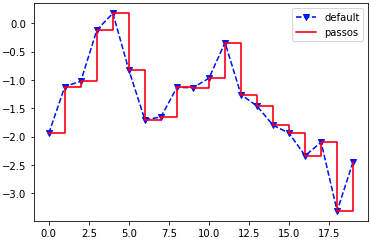
A linha azul é tracejada (‘b–‘), no estilo default e com marcadores ‘v’ (triângulos). O parâmetro label cria legendas, nesse caso indicando o texto ‘defaul’. A linha vermelha tem estilo drawstyle=’steps-post’ (em passos) e é marcada na legenda como ‘passos’. plt.legend(loc='best') informa que o melhor local para colocar essa legenda seja encontrado automaticamente. Outros valores seriam: loc='right', 'center', 'upper right', etc.
Marcas, etiquetas e legendas (ticks, labels, legends)
» dados = np.random.randn(1000) » cumulativo = dados.cumsum() » fig = plt.figure() » ax = fig.add_subplot(1,1,1) » ax.plot(10*dados + 10) » ax.plot(cumulativo)
As duas plotagens são sobrepostas no único axes criado. O operação 10*dados+10 serve apenas para efeito estético da apresentação dos dados aleatórios.
Vamos usar os mesmos dados para verificar as propriedades de ajuste do título global do gráfico, labels nos eixos x e y, label do gráfico e ticks para melhorar a apresentação do gráfico anterior.
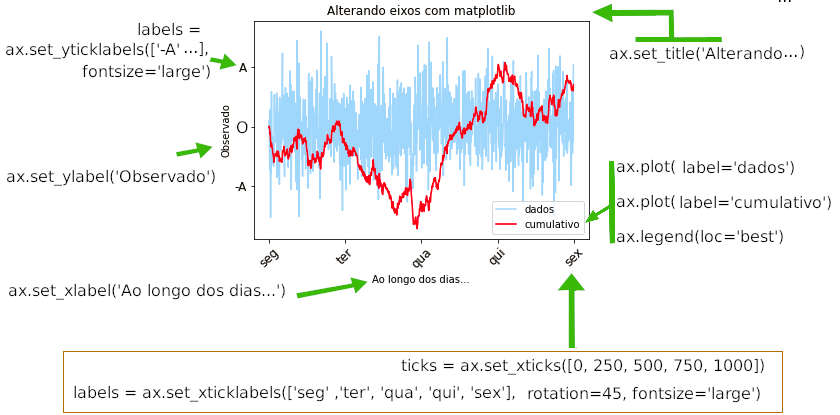
» fig = plt.figure()
» ax = fig.add_subplot(1,1,1)
» ax.set_title('Alterando eixos com matplotlib')
» ticks = ax.set_xticks([0, 250, 500, 750, 1000])
» ticks = ax.set_yticks([-20, 0, 20, 40, 60, 80])
» labels = ax.set_xticklabels(['seg' ,'ter', 'qua', 'qui', 'sex'],
» rotation=45, fontsize='large')
» labels = ax.set_yticklabels(['-A' ,'O', 'A', 'B', 'C','D'],
» fontsize='large')
» ax.set_xlabel('Ao longo dos dias...')
» ax.set_ylabel('Observado')
» ax.plot(10*dados, color='#55aaff', alpha=.5, label='dados')
» ax.plot(cumulativo, color='red', label='cumulativo')
» ax.legend(loc='best')
Anotações e desenhos nos subplots.
Pular para a seção Plotagem em barras.
Diversos tipos de anotações, setas e desenhos podem ser incluídos nos gráficos. Para traçar os gráficos seguintes vamos usar o arquivo .csv do Our World in Data†, baixados para a pasta ./dados.
Primeiro importamos o arquivo baixado .csv para um dataframe. Esse arquivo contém dados dos países do mundo, nos anos de 1950 até 2099, contendo número de nascimentos verificados até 2020 e valores interpolados para os anos seguintes. O dataframe original tem o seguinte formato:
Em seguida selecionamos apenas os dados sobre o Brasil.
» dados = pd.read_csv('./dados/number-of-births-per-year.csv')
» dados=dados[(dados['Entity']=='Brazil')]
» dados.head(2)
» # as colunas 3 e 4 têm nomes longos, que vamos renomear
» dados.columns[3],dados.columns[4]
↳ ('Estimates, 1950 - 2020: Annually interpolated demographic indicators - Births (thousands)',
↳ 'Medium fertility variant, 2020 - 2099: Annually interpolated demographic indicators - Births (thousands)')
» dados.rename(columns={'Year':'ano',
» dados.columns[3]:'nasc',
» dados.columns[4]:'inter'}, inplace=True)
» # copiamos os dados da coluna de interpolação, após 2020, para a colunas de nascimentos
» dados.loc[dados['nasc'].isna(), 'nasc'] = dados['inter']
» # vamos mantes apenas as colunas 'ano' e 'nasc'
» dados = dados[['ano', 'nasc']]
» dados.head()
↳ ano nasc
↳ 4050 1950 2439820.0
↳ 4051 1951 2467186.0
↳ 4052 1952 2523577.0
↳ 4053 1953 2583285.0
↳ 4054 1954 2646311.0
Para usar como anotações no gráfico encontramos os anos em que nascimentos foram máximo e mínimo, além do ano em que se inicia a interpolação, 2020.
» maior=dados[dados['nasc']==dados['nasc'].max()]
» menor=dados[dados['nasc']==dados['nasc'].min()]
» ano_maior = maior['ano'].values[0]
» nasc_maior = int(maior['nasc'].values[0])
» ano_menor = menor['ano'].values[0]
» nasc_menor = int(menor['nasc'].values[0])
» interX = 2020 # início da interpolação
» interY = int(dados[dados['ano']==2020]['nasc'].values[0])
» txt = ('Máximo de nascimentos:\t {} no ano {}.\n'
» 'Mínimo de nascimentos:\t {} no ano {}.\n'
» 'Início da interpolação:\t {} no ano {}.'
» )
» print(txt.format(nasc_maior, ano_maior,nasc_menor, ano_menor, interY, interX))
↳ Máximo de nascimentos: 3929646 no ano 1983.
↳ Mínimo de nascimentos: 1504597 no ano 2099.
↳ Início da interpolação: 2859135 no ano 2020.
Com esses dados imprimimos o gráfico (sem muita preocupação estética). Uma primeira curva é traçada em preto, incluindo os anos de 1950 até 2020. A segunda curve se inicia em 2021 até o final e é tracejada em vermelho, para indicar a interpolação. Uma terceira curva tem efeito decorativo, em azul e transparente.
» fig = plt.figure()
» ax = fig.add_subplot()
» ax.set_title('Nascimentos (em milhões), por ano', size=18)
» ax.plot(dados[dados['ano']<2021]['ano'], dados[dados['ano']<2021]['nasc'], color='black', alpha=1)
» ax.plot(dados[dados['ano']>2020]['ano'], dados[dados['ano']>2020]['nasc'], 'r--')
» ax.plot(dados['ano'], dados['nasc'], 'b', linewidth=5, alpha=.2)
» ax.grid(color='grey', alpha=.3 )
» ax.annotate('Máximo', xy=(ano_maior, nasc_maior), size=13)
» ax.annotate('Mínimo', xy=(ano_menor, nasc_menor), size=13)
» ax.annotate('Interpolado', xy=(interX, interY), size=13)
» ax.arrow(ano_maior-10, nasc_maior, 10, 0)
» ax.arrow(interX-10, interY-10, 10, 10) # a figura 17 abaixo é plotada
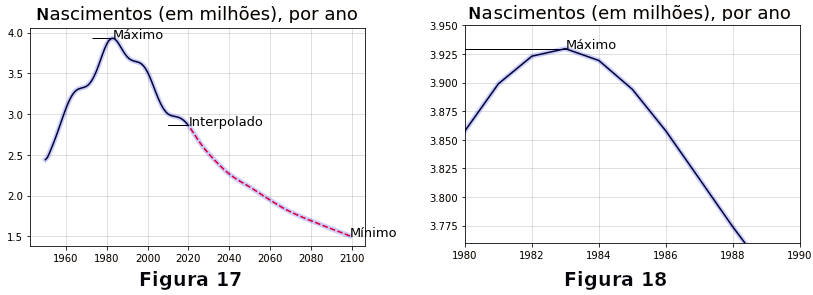
Para dar um zoom na figura podemos limitar as faixas de valores no eixo x e eixo y com ax.set_xlim(a,b) e ax.set_xlim(m,n), respectivamente, em torno do ponto de interesse. No exemplo fazemos um zoom em torno do ponto de máximo, obtendo o gráfico 18 acima.
» ax.set_xlim([1980, 1990]) » ax.set_ylim([3.76e6, 3.95e6]) » fig.get_figure() # a figura 18 acima é plotada
Figuras sobre o plot
Diversas formas mais comuns estão disponíveis para inserção nos plots, e são chamadas de patches no matplotlib. Algumas delas estão diretamente em matplotlib.pyplot como retângulos, círculos e polígonos. Muitas outras estão em matplotlib.patches. Para traçar figuras construimos os patches com os métodos apropriados e os acrescentamos ao subplot usando ax.add_patch().
| retângulo: | plt.Rectangle((x, y), largura, altura), onde (x, y) são as coordenadas do ponto inferior esquerdo, |
| círculo: | plt.Circle((x_0, y_0), raio), onde (x_0, y_0) são as coordenadas do centro, |
| polígono: | plt.Polygon([[x_0, y_0], [x_1>, y_1],…, [x_n, y_n]). |
No caso do polígono a área interna às retas que ligam os pontos é colorida.
» fig = plt.figure() » ax = fig.add_subplot() » retangulo = plt.Rectangle((0.2, 0.2), 0.6, 0.4, color='#aabbcc') » circulo = plt.Circle((0.4, 0.6), 0.3, color='plum', alpha=0.3) » poligono1 = plt.Polygon([[0.1, 0.1], [0.8, 0.7], [.3,.7], [0.6, 0.1]], color='turquoise', alpha=0.8) » poligono2 = plt.Polygon([[0.2, 0.2], [0.8, 0.8]], color='red', alpha=0.8) » ax.add_patch(retangulo) » ax.add_patch(circulo) » ax.add_patch(poligono1) » ax.add_patch(poligono2)
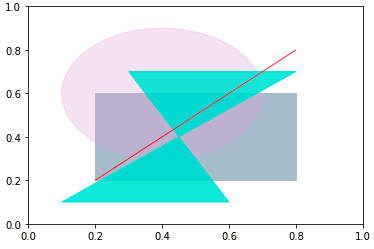
Para o segundo “polígono” apenas dois pontos foram fornecidos e ele é representado pela reta (vermelha) que liga esses pontos.
Claro que gráficos mais elaborados podem ser montados com uma combinação de figuras como retas, polígonos, círculos, etc. No caso abaixo uma cor é escolhida “aleatoriamente” para plotar um série de 2 triângulos, um com um vértice em (0,1), outro em (1,0). Os dois outros vértices dos triângulos são coincidentes, e se deslocam sobre a reta (t,t) com t de 0 a 1, com espaçamento .1. A cor tem transparência alpha=.6 para que as cores apareçam em tons pastéis.
A função cor() retorna uma tupla (r,b,g) onde cada componente representa as cores vermelho, verde e azul, com valores de 0 até 1.
» def cor(): » return (np.random.random(), np.random.random(), np.random.random()) » fig = plt.figure() » ax = fig.add_subplot() » for t in np.linspace(0,1,100): » c = cor() » poligono1 = plt.Polygon([[0, 1], [t, t], [t+.1, t+.1]], color=c, alpha=.6) » poligono2 = plt.Polygon([[1, 0], [t, t], [t+.1, t+.1]], color=c, alpha=.6) » ax.add_patch(poligono1) » ax.add_patch(poligono2) # a figura 20 é gerada
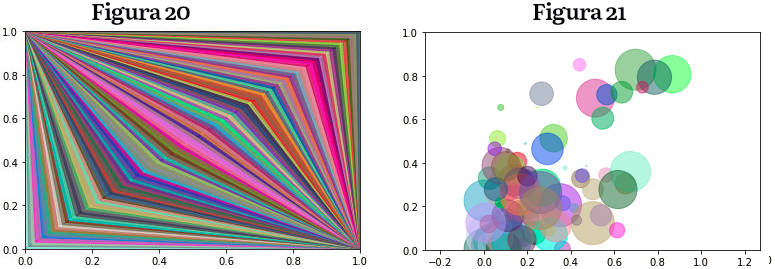
O código abaixo gera círculos de raios aleatórios, espalhados em torno da reta (t,t), afastados dela por uma variacão também aleatória.
» fig = plt.figure()
» ax = fig.add_subplot()
» plt.axis('equal')
» for t in np.linspace(0,1,100):
» circulo = plt.Circle((t*np.random.random(), t*np.random.random()), np.random.random()/10, color=cor(), alpha=0.5)
» ax.add_patch(circulo)
» ax.set_xlim([0, 1])
» ax.set_ylim([0, 1])
» plt.savefig('circulos.pdf') # a figura 21 é gerada
Ao final a figura gerada é gravada em disco com o formato “pdf”. Outros formatos podem ser escolhidos, como “jpeg”, “png”, “svg”, assim como a resolução em dots per inches, (dpi ), que tem default = 100, além da cor de fundo e bordas.
Configuração do matplotlib
Por padrão o matplotlib possui um esquema de cores e outros parâmetros, como largura e tipo de linhas, previamente definidos e voltados para plotar figuras prontas para publicação. No entanto, vários destes parâmetros podem ser personalizados através de ajustes nos valores globais tais como tamanho, espaçamento entre subplots, cores, família e tamanhos de fonte, estilos de grade, etc.
Uma forma de alterar esses padrões está no método plt.rc (parametro, opcoes) onde parametro é uma string com o nome do parâmetro que se quer modificar, e opcoes é uma sequência de argumentos de palavras-chaves com os novos valores.
Entre outras opções parametro pode ser figure, axis, xtick, ytick, grid, legend. As opções podem ser passadas de várias formas. O exemplo mostra como ajustar todas as figuras de uma sessão para o tamanho 20×15. Depois fazemos ajustes às fontes, usando um dicionário.
» # ajustar tamanho da figura
» plt.rc ('figure', figsize = (20, 15))
» # parâmetros associados às fontes, em um dicionário
» font_options = {'family' : 'monospace', 'weight' : 'bold', 'size' : 'small'}
» plt.rc ('font', **font_options)
Uma personalização mais ampla pode ser feita no arquivo de configurações. Para encontrar esse arquivo use os comandos:
» # no prompt do sistema digite » python -c "import matplotlib; print(matplotlib.matplotlib_fname())" » # de dentro do jupyter notebook (ou de qualquer ambiente em que você trabalhe): » import matplotlib » print(matplotlib.matplotlib_fname()) ↳ /home/usuario/.anaconda3/lib/python3.8/site-packages/matplotlib/mpl-data/matplotlibrc
Esse output é relativo ao sistema e à distribuição que está sendo usada, lembrando que é possível existir mais de uma instalação em um computador. No caso mostrado está em uso o anaconda e jupyter no linux mint. Cada usuário pode encontrar um local diferente. Esse arquivo não deve ser editado diretamente mas copiado para a pasta home do usuário (no linux) com o nome .matplotlibrc. Desta forma ele será carregado durante a inicialização do pacote. A análise desse arquivo é uma boa forma de se conhecer as possibilidades na personalização, sendo que as opções estão comentadas.
As atuais configurações globais podem ser vistas com o comando
» import matplotlib as mpl » print(mpl.rcParams)
matplotlib.rcParams é usado para alterar esses parâmetros, um de cada vez. matplotlib.rc pode alterar os valores default para vários parâmetros de um grupo específico, como tipos de lihnes, fontes, textos, etc.
» matplotlib.rcParams['lines.markersize'] = 20 » matplotlib.rcParams['font.size'] = '15.0'
matplotlib.rcdefaults() reseta todos os parâmetros para seus valores originais.
Usando matplotlib com o pandas
A própria biblioteca do pandas embute diversas funcionalidades do matplotlib, sem que esse tenha que ser carregado explicitamente. Isso significa que podemos criar gráficos sem passar por todas as etapas de sua construção.
Por exemplo, uma instância de Series possui o método series.plot().
» s1 = pd.Series(np.random.randn(100).cumsum()) » s1.plot(use_index=False) # grafico 22-a é plotado » s2 = pd.Series([x**2 for x in np.arange(-10,10,.1)], index=np.arange(-10,10,.1)) » s2.plot() # grafico 22-b é plotado
Na figura 21 o eixo x recebe valores dos índices da série, que por default vai de 0 até 99. Na segunda o índice que foi declarado é usado. Para evitar o procedimento de usar o índice como ordenada passamos o parâmetro series.plot(use_index=False).
Para um dataframe cada series correspondente a cada coluna é plotada separadamente. Abaixo construimos e plotamos um dataframe de quatro colunas, cada uma delas representando valores de um seno com frequências diferentes.
» s1 = pd.Series([np.sin(x) for x in np.arange(0,10,.1)])
» s2 = pd.Series([np.sin(2*x) for x in np.arange(0,10,.1)])
» s3 = pd.Series([np.sin(3*x) for x in np.arange(0,10,.1)])
» s4 = pd.Series([np.sin(4*x) for x in np.arange(0,10,.1)])
» df=pd.concat({'A': s1, 'B': s2, 'C': s3, 'D': s4} , axis=1)
» df.plot() # a figura 23 é plotada
» df.plot(color=['k','r','b','y'], alpha=.6, logx=True, grid=True) # a figura 24 é plotada
O dataframe df contém 4 colunas, cada uma com os valores de seno(πx), seno(2πx), seno(3πx), seno(4πx), com x variando de 0 a 10 em passos de 0,1. No segundo gráfico, figura 24, alguns parâmetros foram passados, como uma lista de cores, a existência de quadriculado (grid) e a instrução para usar uma escala logarítmica em x.
A instrução dataframe.plot() é um atalho para dataframe.plot.line() que representa como curvas os pontos passados. Outros parâmetros podem ser passados, exatamente como no uso direto de matplotlib:
| Argumento | Descrição |
|---|---|
| label | texto para a legenda |
| ax | objeto subplot do matplotlib onde plotar. Se vazio os plots vão para o subplot ativo |
| style | string de estilo, como ‘ko–‘, passado para o matplotlib |
| alpha | opacidade do plot (de 0 to 1) |
| kind | opções: ‘area’, ‘bar’, ‘barh’, ‘density’, ‘hist’, ‘kde’, ‘line’, ‘pie’ |
| logy | use escala logaritmica no eixo y |
| use_index | use o index para os labels de x |
| rot | rotação de texto nos labels (0 até 360) |
| xticks | valores a usar para marcas no eixo x |
| yticks | valores a usar para marcas no eixo y |
| xlim | limites para o eixo x (ex.: [0, 10]) |
| ylim | limites para o eixo y |
| grid | exibir grade quadriculada de fundo (grid), default=exibir |
Alguns parâmetros são específicos para dataframes.
| Argumento | Descrição |
|---|---|
| subplots | bool. Plota cada coluna em um subplot separado |
| sharex | se subplots=True, use o mesmo eixo x, com marcas e limites |
| sharey | se subplots=True, use o mesmo eixo y, com marcas e limites |
| figsize | tupla indicando tamanho da figura |
| title | texto para o título |
| legend | bool. Inclui legenda do subplot (default=True) |
| sort_columns | plot colunas em ordem alfabética no nome; default= ordem no dataframe |
Plotagem em barras
A plotagem em barras (bar plots ) pode ser feita com plot.bar() e plot.barh() (com barras verticais e horizontais).
Para experimentar com esses tipos de plotagens vamos usar os mesmos dados importados anteriormente, que contém uma lista de países com o número de nascimentos por ano de 1950 até 2020, e a estimativa à partir de 2021. Importamos o arquivo .csv para um dataframe e selecionamos apenas as linhas relativas ao ano de 2020. Linhas relativas à continentes e outras partes do mundo que não países possuem coluna Code = NaN e são excluídas. Renomeamos as colunas para mais fácil manuseio e mantemos apenas as colunas relativas ao país, ano e número de nascimentos.
» # importação do csv em um dataframe
» dados = pd.read_csv('./dados/number-of-births-per-year.csv')
» # selecão do ano = 2020 e apenas países
» dados = dados[(dados['Code'].notnull()) & (dados['Year']==2020)]
» # renomeando colunas
» dados = dados.rename(columns={'Entity':'país','Year':'ano', dados.columns[3]:'nasc'})
» # mantendo apenas colunas relevantes
» dados = dados[['país', 'nasc']]
» # use a coluna 'país' como índice
» dados.set_index('país', inplace=True)
» # o dataframe final:
» dados.head(4)
↳ nasc
↳ país
↳ Afghanistan 1215628.0
↳ Albania 32888.0
↳ Algeria 995368.0
↳ Angola 1311356.0
Imprimimos os 2 tipos de barplot com o código abaixo, usando 10 países para as barras verticais e 20 para as horizontais. Para isso inicializamos uma figura com 1 linha e 2 colunas. O parâmetro figsize=(15, 16) indica que nossa figura terá a largura 15 e altura 6 (em polegadas). Dados os nomes longos de países o gráfico ficou sobreposto, o que seria controlado aumentando-se a separação entre axes.
» fig, axes = plt.subplots(2, 1, figsize=(10, 10)) » dados[:10].plot.bar(ax=axes[0], color=['r','b','g'], rot=30, grid=True) » dados[:20].plot.barh(ax=axes[1], color='g', alpha=0.7, grid=True)
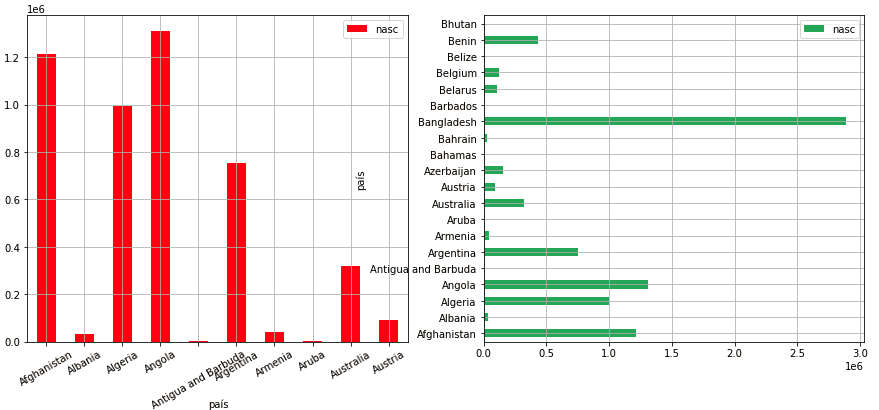
Gráficos desse tipo são desenhados para series e dataframes com apenas uma coluna. Se o dataframe possui várias colunas o gráfico de barras plota uma barra para cada coluna.
Para o próximo exemplo usaremos os dados disponibilizados no GapMinder, já usados e descritos nesse site. Desses dados manteremos apenas as colunas que renomearemos como “pais”, “ano” e “pop” (população), ficando com um dataframe com os países de mundo e suas populações nos anos listados abaixo.
Dessa coleta de dado separamos apenas os 5 países com maior população no último ano, 2007.
» # baixar dados do gapminder
» url =(
» 'https://raw.githubusercontent.com/jennybc/'
» 'gapminder/master/data-raw/08_gap-every-five-years.tsv'
» )
» # criamos o dataframe dfPaises. O arquivo importado tem campos separados por tabs
» dfPaises = pd.read_csv(url, sep='\t')
» # para ver colunas e forma geral usamos
» dfPaises.head(2)
↳ country continent year lifeExp pop gdpPercap
↳ 0 Afghanistan Asia 1952 28.801 8425333 779.445314
↳ 1 Afghanistan Asia 1957 30.332 9240934 820.853030
» # usamos apenas 3 colunas
» dfPaises = dfPaises[['country','year','pop']]
» # e as renomeamos
» dfPaises.rename(columns={'country':'pais', 'year':'ano'}, inplace=True)
» # o resultado é
» dfPaises
↳ pais ano pop
↳ 0 Afghanistan 1952 8425333
↳ 1 Afghanistan 1957 9240934
↳ 2 Afghanistan 1962 10267083
» # para ver os anos registrados examinamos o conjunto (set)
» set(dfPaises['ano'])
↳ {1952, 1957, 1962, 1967, 1972, 1977, 1982, 1987, 1992, 1997, 2002, 2007}
» # países mais populosos, em 2007
» dfPaises[dfPaises['ano']==2007] \
» .sort_values(by=['pop'], axis=0, \
» ascending=False, inplace=False)['pais'] \
» .head(5)
↳ 299 China
↳ 707 India
↳ 1619 United States
↳ 719 Indonesia
↳ 179 Brazil
O último comando, para selecionar os países mais populosos, está quebrada pelo caracter \ (back slash) que é ignorado (a linha é executada por inteiro). Essa linha pode ser compreendida assim:
dfPaises[dfPaises['ano']==2007] : seleção só de linhas com ano = 2007 .sort_values(by=['pop'], axis=0, ascending=False) : ordena pela coluna 'pop' em ordem inversa ['pais'].head(5) : só a coluna 'pais', 5 primeiros valores
Em seguida montamos um dataframe para cada desses países e os concatenamos para um dataframe mais geral que contém linhas indexadas pela ano e colunas com o nome do país. (Outras técnicas de agrupamento serão vistas mais tarde.)
» china = dfPaises[dfPaises['pais']=='China'][['ano', 'pop']]\
» .set_index('ano').rename(columns={'pop':'china'})
» india = dfPaises[dfPaises['pais']=='India'][['ano', 'pop']]\
» .set_index('ano').rename(columns={'pop':'india'})
» usa = dfPaises[dfPaises['pais']=='United States'][['ano', 'pop']]\
» .set_index('ano').rename(columns={'pop':'usa'})
» indonesia = dfPaises[dfPaises['pais']=='Indonesia'][['ano', 'pop']]\
» .set_index('ano').rename(columns={'pop':'indonesia'})
» brasil = dfPaises[dfPaises['pais']=='Brazil'][['ano', 'pop']]\
» .set_index('ano').rename(columns={'pop':'brasil'})
As linhas de seleção de dados do país podem ser compreendidas assim:
dfPaises[dfPaises['pais']=='Brazil'] : seleciona apenas linhas relativas ao país 'Brazil'
[['ano', 'pop']] : desse df copia apenas as colunas 'ano' e 'pop'
.set_index('ano') : use a coluna 'ano' como índice
.rename(columns={'pop':'brasil'}) : renomeie coluna 'pop' para 'brasil'
» # os paises são concatenados em um único df » df = pd.concat([china, india, usa, indonesia, brasil], axis=1) » # o nome da lista de colunas será usado no plot » df.columns.name = 'População' » # e o resultado é » df.head(3) ↳ População china india usa indonesia brasil ↳ ano ↳ 1952 556263527 372000000 157553000 82052000 56602560 ↳ 1957 637408000 409000000 171984000 90124000 65551171 ↳ 1962 665770000 454000000 186538000 99028000 76039390 » # esse dataframe pode ser exibido em gráfico de barras » cor = ['salmon','gold','teal','plum','powderblue'] » df.plot.bar(figsize=(10,5), grid=True, color = cor, title='Países mais populosos (de 1952 até 2007)', rot=45)
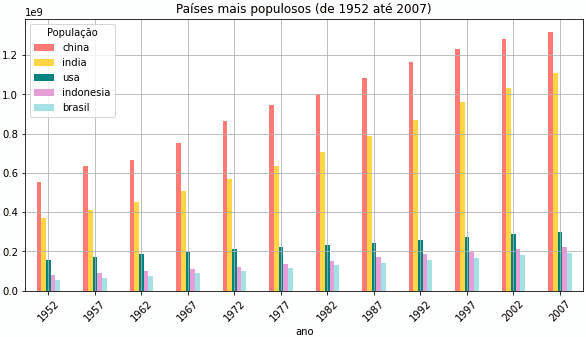
Na plotagem acima uma paleta de cores foi passada para o parâmetro color. Cada uma delas é usada para um país. O nome das colunas, df.columns.name = 'População' é usado como título da legenda.
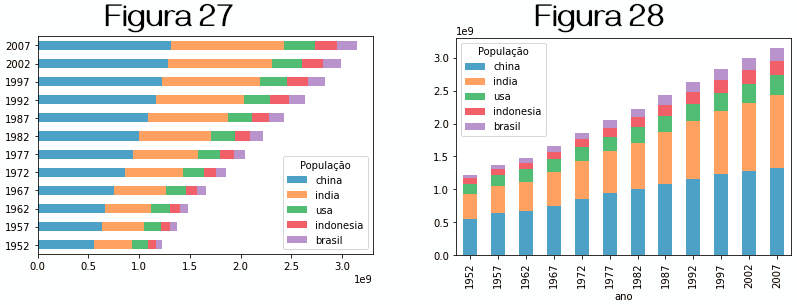
Para gerar gráficos de barras empilhadas (stacked bar ) passamos o valor stacked=True. Nos exemplos plotamos a versão horizontal e vertical do mesmo gráfico acima.
» df.plot.barh(stacked=True, alpha=.7) # a figura 27 é plotada » df.plot.bar(stacked=True, alpha=.7) # a figura 28 é plotada
Histogramas
Um histograma é uma representação gráfica, similar a um gráfico de barras, de uma distribuição de pontos. Os pontos são distribuídos em faixas igualmente divididas e o gráfico é o conjunto de retângulos com base de tamanho igual à largura das faixas e altura correspondente ao número de pontos em cada faixa.
Criamos uma série com 1000 números aleatórios, multiplicados por 100. O resultado é, no caso dessa execução, um conjunto distribuído entre -315 e 325 (aproximadamente). A partir desses dados traçamos o histograma e o gráfico de densidade ou density plot.
» ser = pd.Series((np.random.randn(1000)*100)) » ser.describe() ↳ count 1000.000000 ↳ mean 3.747373 ↳ std 102.637489 ↳ min -314.443835 ↳ 25% -65.343082 ↳ 50% 4.835384 ↳ 75% 73.314604 ↳ max 324.011000 » ser.plot.hist(bins=10, grid=True, color='b', alpha=.4) # figura 19 » ser.plot.kde() # o mesmo que ser.plot.density() # figura 30
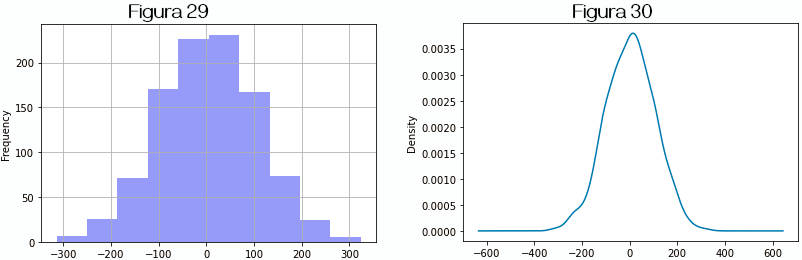
O gráfico de densidade consiste na plotagem de uma função de distribuição de probabilidade que poderia ter gerado os dados na series. A técnica usual consiste em usar uma mistura de “núcleos” ou “kernels”. Esses gráficos são também chamados de estimativa de núcleos de densidade (kernel density estimate, KDE ).
Seaborn
Seaborn é outra biblioteca do Python voltada para a visualização de dados, baseada no matplotlib. Ela apresenta uma interface de mais alto nível e aprimoramento da qualidade estética dos gráficos. Com o Seaborn se pode conseguir gráficos bem elaborados e de boa aparência com um número menor de linhas de código.
Para os exemplos com o seaborn vamos usar os dados do Gapminder já descritos. O dataframe importado tem 1704 linhas com dados sobre os países, 6 colunas ‘country’, ‘continent’, ‘year’, ‘lifeExp’, ‘pop’, ‘gdpPercap’, respectivamente ‘pais’, ‘continente’, ‘ano’, ‘Expectativa de vida’, ‘população’, ‘PIB percapita’.
» url =( » 'https://raw.githubusercontent.com/jennybc/' » 'gapminder/master/data-raw/08_gap-every-five-years.tsv' » ) » # criamos o dataframe dfPaises. O arquivo importado tem campos separados por tabs » dfPaises = pd.read_csv(url, sep='\t') » # para restringir o volume dos dados armazenamos as fatias » df2007 = dfPaises[dfPaises['year']==2007] » dfBrasil = dfPaises[dfPaises['country']=='Brazil']
O seaborn deve ser importado. Para uma gráfico de barras mais simples informamos e base de dados e os nomes de colunas a usadas como valores para os eixos. Para não congestionar o gráfico usamos apenas os 10 primeiros países.
» import seaborn as sns » sns.barplot(data=df2007[:10], x='lifeExp', y='gdpPercap') » # é plotada a figura 31 abaixo
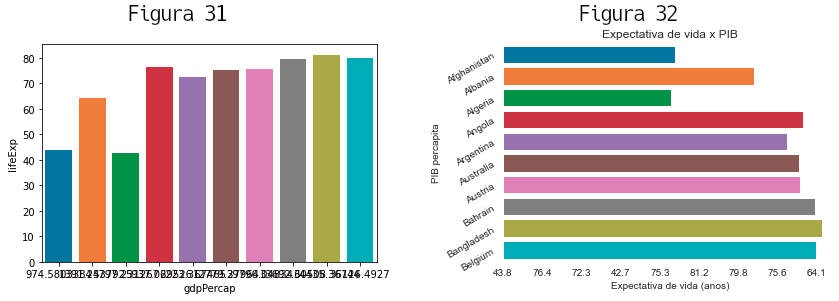
Muitas configuarações podem ser aplicadas sobre esse gráfico básico. Algumas são usadas abaixo, como a orientação das barras, textos e rotação nos eixos x e y.
» sns.set_style('darkgrid')
» graf = sns.barplot(data=df2007[:10], x='lifeExp', y='gdpPercap', orient='h')
» graf.set(xlabel = 'Expectativa de vida (anos)',
ylabel = 'PIB percapita', title ='Expectativa de vida x PIB')
» graf.set_xticklabels(labels=df2007[:10]['lifeExp'].round(1))
» graf.set_yticklabels(labels=df2007[:10]['country'], rotation=30)
» # é plotada a figura 32 acima
Valores válidos para sns.set_style() são 'white', 'dark', 'whitegrid', 'darkgrid', 'ticks'.
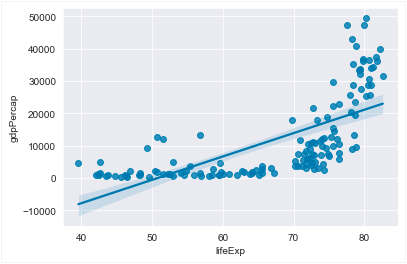
Esses gráficos plotados não sugerem qualquer relação entre a renda percapita e a expectativa de vida, o que é natural uma vez que escolhemos apenas os primeiros 10 países, em ordem alfabética. Claro que barplots não são apropriados para exibir um número muito grande de dados. Para isso podemos usar seaborn.regplot que plota o gráficos de dispersão (scatter plots ) e uma reta correspondente a um ajuste do modelo de regressão linear. Esse último gráfico mostra que existe correlação entre expectativa de vida e renda percapita.
» sns.regplot(x='lifeExp', y='gdpPercap', data=df2007) # figura 33
É comum em análise de dados que se queira ter uma visão geral de relacionamentos entre as variáveis (ou colunas de um dataframe. Para isso um pairplot faz o cruzamento entre todas as variáveis. O método seaborn.pairplot(), por default, cria uma matriz de Axes comparando aos pares as variáveis numéricas do dataframe usado como fonte de dados. Na diagonal dessa matriz uma distribuição univariada é exibida para mostrar a distribuição dos dados em cada coluna.
» sns.pairplot(df2007[['gdpPercap', 'lifeExp']],
» diag_kind='kde', plot_kws={'color':'r','alpha': .9})
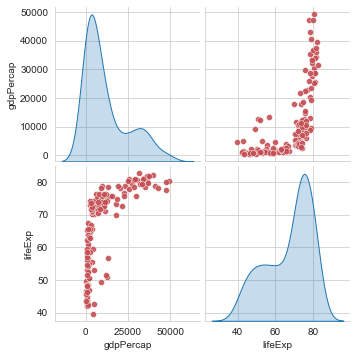
O parâmetro plot_kws recebe um dicionário de propriedades com valores.
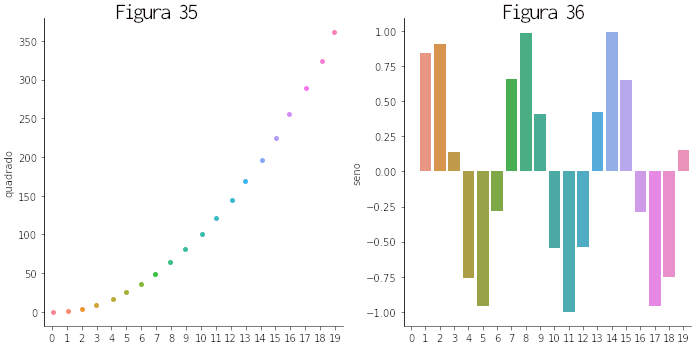
Outro método é seaborn.catplot() que traz diversas funcionalidades para representar relações entre variáveis numéricas ou categóricas. Para uma amostra criamos um dataframe com valores de uma parábola e um seno.
» dfGraf=pd.DataFrame(np.arange(20), columns = ['x']) » dfGraf['quadrado']=dfGraf['x']**2 » dfGraf['seno']=np.sin(dfGraf['x']) » sns.catplot(x='x', y='quadrado', kind='strip', data=dfGraf) # plota a figura 35 » sns.catplot(x='x', y='seno', kind='bar', data=dfGraf) # plota a figura 36
Bibliografia
- McKinney, Wes: Python for Data Analysis, Data Wrangling with Pandas, NumPy,and IPython
O’Reilly Media, 2018. - Poladi, Srinivasa R.: Matplotlib 3.0 Cookbook, Packt, Mumbai, 2018.
- Realpython: matplotlib guide.
- Matplotlib.org Matplotlib.
- Site Seaborn: Seaborn.
- Site Towards Data Science Mastering catplot() in Seaborn: Categorical data visualization guide..
Todos os sites acessados em setembro de 2021.
Consulte bibliografia completa em Pandas, Introdução neste site.





