Gráficos com plot
Uma parte importante da análise de dados esta na visualização destes dados em forma gráfica. A representação visual de dados permite, muitas vêzes, o reconhecimento de padrões que dificilmente seriam percebidos apenas com tabelas e números. R fornece várias funções para representar dados graficamente, tanto em gráficos bidimensionais quanto tridimensionais. Em particular dá-se ênfase aos gráficos estatísticos, tais como histogramas, curvas de distribuições, gráfico de barras e outros. Existem métodos gerais que se aplicam à diversas formas básicas de gráficos. Pode-se incluir títulos, nomes para os eixos, cores, representações por pontos linhas e sinais variados e anotações.
A função plot() é a mais básica na geração de um gráfico.
> x <- -10:10; y <- x^2; plot(x,y) # resultado na figura 1 > # Parâmetro para tornar a linha contínua > plot(x,y, type="l") # resultado na figura 2

Alguns dos parâmetros são listados abaixo:
plot(x, y, ...)
| x | coordenadas horizontais dos pontos. Pode ser um objeto numerado. |
| y | coordenadas verticais dos pontos. Omitido se x é um objeto numerado. |
| … | Argumentos adicionais (parâmetros gráficos). |
Entre os argumentos adicionais:
| type = | “p” (pontos), “l” (linhas), “b” (ambos), |
| “h” (histograma), “s” (degraus), “n” (em branco) | |
| main = | título principal |
| sub = | subtítulo |
| xlab = | título para eixo x |
| ylab = | título para eixo y |
| asp = | aspecto (razão y/x). |
Observe que, se u é um objeto ordenado, então plot usa como coordenada x a ordem dos elementos. Por exemplo:
> u <- rnorm(10) > # Os dois gráficos seguintes são idênticos > plot(u) > plot(1:10, u)
Algumas funções sobreescrevem o gráfico já traçado. É o caso da função lines. É possível alterar características das linhas com os parâmetros lwd (largura da linha) e lty (tipo da linha), como se mostra no exemplo:
> a <- 1:20; b <- a^2 > plot(a, .1*b ,type="l") # linha 1 > lines(a, .2*b , lwd=4 ) # linha 2 > lines(a, .3*b , lwd=2) # linha 3 > lines(a, .4*b , lty=3) # linha 4 > points(a,.5*b, pch=3) # pontos 5 > text(10, 2, "Título do gráfico") # título na posição 10 , 2
As linhas acima resultam no gráfico:

Se vários gráficos devem ser analisados ao mesmo tempo, uma nova instância da janela de saída gráfica pode ser aberta com o comando dev.new() ou X11() (apenas para sistemas tipo Unix). Pode-se navegar entre “devices” gráficos abertos usando dev.new(), dev.next(), dev.prev(), dev.set() e dev.off().
> plot(rnorm(10)) # plota o primeiro gráfico > dev.new() # abre nova janela > plot(rnorm(20)) # plota o segundo gráfico
Os gráficos podem ser enviados diretamente para arquivos nos formatos JPEG, BMP, PDF, TIFF, PNG, entre outros. Como exemplo, direcionamos a saída gráfica para um arquivo JPEG:
> # grava arquivo hiperbole.jpg > jpeg(file='hiperbole.jpg') > # plota gráfico > plot(x<- -100:100, 1/x, type='l', main="hipérbole") > # fecha janela gráfica > dev.off() > # Grava arquivo jpeg com a imagem à direita.
Outras funções capazes de escrever por cima de um gráfico já plotado são locator(n) e identify(). A primeira serve para que o se selecione regiões do gráfico utilizando o botão esquerdo do mouse até que se tenha um número n de pontos selecionados (ou até pressionar o botão direito do mouse, para terminar). A cada clique dado com o botão esquerdo do mouse a função retorna no console as coordenadas do clique. Por exemplo:
> x <- 1:100; y <- sqrt(x) > plot(x,y, type="l") > text(locator(1), "x é aqui!") > text(locator(1), "y é aqui!") > text(locator(5), paste("<", 1:4, ">") > # A linha acima marca 4 pontos > # no gráfico, com o texto: > # "<1>", "<2>", "<3>", "<4>" > # respectivamente
Para os próximos passos usaremos o dataframe carregado por padrão no R de nome mtcars. Este é um conjunto de dados sobre automóveis com campos mpg, cyl, disp, hp, drat, wt, qsec, vs, am, gear, carb. Antes de prosseguir, observe que as quatro formas de notação abaixo são equivalentes:
> # ------------------------------ (1) > plot( mtcars$mpg, mtcars$cyl) > # ------------------------------ (2) > attach(mtcars) > plot( mpg, cyl) > detach(mtcars) > # ------------------------------ (3) > attach(mtcars) > plot(cyl ~ mpg) > detach(mtcars) > # ------------------------------ (4) > plot( cyl ~ mpg, data= mtcars )
Na quarta forma usamos y ~ x para representar x como variável independente, y como variável dependente.
Aproveitamos a oportunidade para conhecer uma sintaxe especial. O comando plot( cyl ~ ., data= mtcars ) realiza a plotagem de todos os gráficos de cyl como função de todos os demais campos de mtcars. O prompt Hit to see next plot: aparece entre as operações.
> attach(mtcars) > plot(mpg ~ wt) > abline(lm(mpg ~ wt)) > title("Regressão Linear de Consumo por Peso") > detach(mtcars)
pdf basta cercar todas as linhas acima pelos comandos pdf("NomeArquivo.pdf") e dev.off().A função abline(a,b) traça uma reta sobre o gráfico, recebendo os parâmetros a como a interseção da reta com o eixo y (x = 0) e b como a inclinação da reta. Ela recebe como argumento lm(y~x) que retorna diversos dados sobre a regressão linear da função y~x, entre eles os parâmetros a e b necessários para definar a reta.
A função abline possui outros parâmetros. Entre eles:
abline(a=NULL, b=NULL, h=NULL, v=NULL, col=, ...)
a = interseção com eixo y
b = inclinação da reta
h = y (traça reta horizontal por y)
v = x (traça reta vertical por x)
col = cor: "red", "blue", etc; ou rgb(x,y,z), onde x, y, z ∈ [0, 1]
ou hexadecimal #abcdef; a, ..., f ∈ [0, f], hexadecimal.
Alguns exemplos de uso de abline(). O código seguinte gera os gráficos abaixo:
> plot(x<- 0:10, x) # plotar uma reta
> title("Reta y = x")
> abline(v=4) # reta vertical por x = 4
> abline(v=6, col="blue") # reta vertical por x = 6, azul
> abline(h=8, col="#779900") # outra cor
> # Usando a tabela cars (embutida em R)
> dev.new()
> plot(cars)
> abline(v=c(15,20), col=c("blue", "red"), lty=c(1,2), lwd=c(1, 3), h=40)

Parâmetros Gráficos
O conjunto de parâmetros para construção de gráficos podem ser lidos e (alguns deles) alterados através da função par(). Podem ser características como fonts, cores, eixos e títulos.
par(..., no.readonly = FALSE)
Argumentos:
no.readonly = Booleano. Se TRUE apenas parâmetros que podem ser
alterados pelo usuário são exibidos.
... Outros parâmetros são passados na forma:
par1 = valor1, ..., parn = valorn
Se nenhum parâmetro for fornecido par() exibe uma lista dos parâmetros atuais, par(no.readonly = TRUE) exibe uma lista dos parâmetros que podem ser alterados. Valores alterados dessa forma permanecem válidos durante a sessão.
Para exemplificar suponha que pretendemos ter nossos gráficos plotados com quadrados sólidos ligados por retas. O seguinte código pode ser usado:
> # parâmetros default são armazenados
> oldPar <- par(no.readonly=TRUE)
> par(lty=1, pch=15)
> plot(cars$dist ~ cars$speed, type="b")
> title("Usando quadrados e retas")
> # parâmetros default são restaurados
> par(oldPar)
> # Claro que o mesmo efeito seria obtido com
> plot(cars$dist ~ cars$speed, type="b", lty=1, pch=15)

Alguns parâmetros são listados na tabela:
| Parâmetro | Descrição |
|---|---|
| pch | símbolo usado para marcar pontos. |
| cex | tamanho do símbolo, relativo ao default. 1 = default, 1.5 is 50% maior, etc. |
| lty | tipo da linha. |
| lwd | largura da linha, relativa ao default. Ex.: lwd=2 dupla largura. |
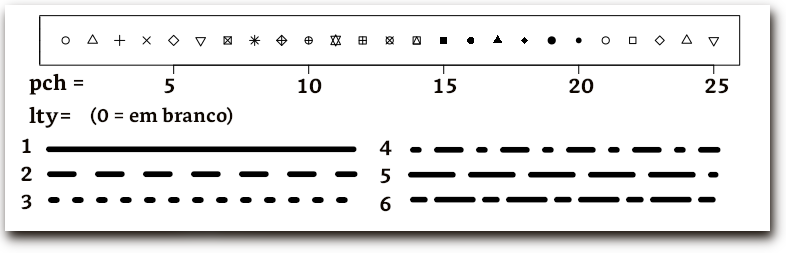
pch, ltyAs cores que podem ser alteradas nos gráficos estão listas na tabela seguinte:
| Parâmetro | Descrição |
|---|---|
| col | cor default do gráfico. |
| col.axis | cor para texto nos eixos. |
| col.lab | cor para labels nos eixos. |
| col.main | cor do título. |
| col.sub | cor do subtítulo. |
| fg | cor do primeiro plano. |
| bg | cor de fundo. |
Para o parâmetro col algumas funções aceitam valores reciclados. Por ex., se col=c("blue", "green") e três curvas são exibidas então a primeira e a terceira serão azuis, a segunda verde. Cores podem ser especificadas por índice, nome, valores hexadecimais, RGB e HSV. A função colors() exibe uma lista de todas as cores disponíveis, por nome.
Algumas funções permitem a criação de palhetas, vetores com n cores contíguas:
rainbow(n, s = 1, v = 1, start = 0, end = max(1, n - 1)/n, alpha = 1,
heat.colors(n, alpha = 1),
terrain.colors(n, alpha = 1),
topo.colors(n, alpha = 1),
cm.colors(n, alpha = 1))
| n | número (≥ 1) de cores no vetor. |
| s, v | “saturação” e “valor” no formato HSV. |
| start | [0,1] cor inicial para o arco-íris (rainbow). |
| end | [0,1] cor final para o arco-íris (rainbow). |
| alpha | [0,1], transparência. |
Vamos explorar o uso destas palhetas de cores na próxima seção.
Para especificar o estilo, tamanho e família das fontes os seguintes parâmetros gráficos podem ser usados:
| Parâmetro | Descrição |
|---|---|
| cex | magnificação do texto: 1 = default, 1.5 = 50% maior; 0.5 = 50% menor, etc. |
| cex.axis | magnificação dos eixos, relativo a cex. |
| cex.lab | magnificação do texto nos eixos, relativo a cex. |
| cex.main | magnificação do texto do título, relativo a cex. |
| cex.main | magnificação do texto do subtítulo, relativo a cex. |
| font | inteiro para fonte: 1 = simples, 2 = negrito, 3 = itálico, 4 = negrito itálico, 5=símbolo. |
| font.axis | fonte nos eixos. |
| font.lab | fonte nos labels de eixos. |
| font.main | fonte nos títulos. |
| font.sub | fonte nos subtítulos. |
| ps | tamanho do ponto na fonte (~1/72 polegada). |
| family | família da fonte. Os padrões são serif, sans e mono |
Por exemplo, após a aplicação dos parâmetros:
par(cex.main=3, cex.lab=1.5, font.lab=2, font.main=4, font.sub=3)
o texto nos gráficos serão representados com: títulos com fontes 3 vezes maior que o padrão definido em cex, eixos magnificados em 1.5, labels em negrito nos eixos, títulos em negrito itálico e subtítulos em itálico.
Para controle das dimensões do gráfico e margens usamos:
| Parâmetro | Descrição |
|---|---|
| pin | largura e altura do gráfico, em polegadas. |
| mai | vetor com larguras das margens, c(inferior, esquerda, superior, direita) em polegadas. |
| mai | vetor com larguras das margens, c(inferior, esquerda, superior, direita) em linhas. [default = c(5, 4, 4, 2) + 0.1]. |
Função barplot()
A função barplot() permite a exibição de gráficos de barras. Um resumo de seus parâmetros está mostrado abaixo.
barplot(height, width = 1, space = NULL, names.arg = NULL,
horiz = FALSE, density = NULL, col = NULL, border = par("fg"),
main = NULL, sub = NULL, xlab = NULL, ylab = NULL, axes = TRUE)
| height | vetor ou matriz contendo altura das barras. |
| width | vetor com largura das barras. |
| space | espaço deixado antes da barras (uma fração da largura). |
| names.arg | vetor de nomes para barras. |
| horiz | booleano. FALSE = barras verticais; TRUE = barras horizontais. |
| density | vetor, densidade do hachurado. NULL= sem hachura. |
| col | vetor de cores das barras. |
| border | cor das bordas das barras. |
| main,sub | título e subtítulo |
| xlab | texto para o eixo x. |
| ylab | texto para o eixo y. |
| axes | booleano. Se eixos são desenhados |
O código acima gera o gráfico de barras abaixo:
A função bar plot pode receber uma matriz como argumento. Para ilustrar vamos usar a função table() para tabelar dados no data frame mtcars. Este data frame possui o campo mtcars$carbs que lista o número de carburadores de uma lista de automóveis. Em seguida criamos uma tabela com um teste clínico hipotético para o tratamento da gripe usando um antiviral, vitammina C e um “chazinho”.
> carburadores <- table(mtcars$carb)
> carburadores
1 2 3 4 6 8
7 10 3 10 1 1
> # A tabela mostra que existem 7 modelos com 1 carburador, 10 com 2, etc.
> barplot(carburadores, main="Modelos x carburadores", horiz=TRUE,
names.arg=c("1", "2", "3","4", "6", "8"), xlab="Quantos modelos",
ylab="Número de carburadores", col=rainbow(6))
> testeClinico <- matrix(c(45,9,12,4,31,31,1,10,7), ncol=3, byrow=TRUE)
> cores <-c("#5FC0A0", "#DE7A6B", "#6BA0DE")
> colnames(testeClinico) <- c("Antiviral","Vitamina C","Chazinho")
> rownames(testeClinico) <- c("Melhorou","Sem alteração","Piorou")
> testeClinico
Antiviral Vitamina C Chazinho
Melhorou 45 9 12
Sem alteração 4 31 31
Piorou 1 10 7
> resultado <- as.table(testeClinico)
> barplot(resultado, main="Gripe: teste clínico", xlab="Medicamento",
ylab="Eficácia", col=cores, legend=rownames(resultado))
O código acima gera os gráficos:

O mesmo gráfico, com os dados agrupados por tipo de medicamento testado pode ser obtido ao se acrescentar o parâmetro beside=TRUE que força a exibição de dados lado à lado para uma mesma coluna:
cores <-c("#5FC0A0", "#DE7A6B", "#6BA0DE") > barplot(resultado, main="Gripe: teste clínico", xlab="Medicamento", ylab="Eficácia", col=cores, beside=TRUE)
Usando funções de agregamento e passando o resultado para barplot() pode-se representar médias, medianas, desvios padrões e outros em gráficos de barras.
Para experimentar com esta funcionalidade usaremos o dataset embutido com o R denomidado states (US State Facts and Figures). Ele contém dados antigos sobre os 50 estados americanos. Em particular usaremos state.region, um fator contendo as regiões de cada estado (Northeast, South, North Central, West) e state.x77, uma matriz com 50 linhas e 8 colunas com informações sobre os estados. O campo state.x77$Illiteracy contém taxas de analfabetismo nos estados americanos em 1970, como porcentagem da população.
> # Carregamos uma palheta de 4 cores
> cor <- c("#F3E16E", "#6EC6F3", "#6FF36E", "#F36E84")
> reg <- state.region
> levels(reg) # as regiões estão em inglês
[1] "Northeast" "South" "North Central" "West"
# Para traduzir para o português alteramos os levels:
> levels(reg) <- c("Nordeste","Sul","Central Norte","Oeste")
> levels(reg)
[1] "Nordeste" "Sul" "Central Norte" "Oeste"
> # Usamos apenas a 3a. coluna de state.x77 (analfabetismo %)
> analfabetismo <- state.x77[,3]
> # Criamos um dataframe com informações: regiões x analfabetismo
> estados <- data.frame(reg, analfabetismo)
> # Agregamos a informação sobre analfabetismo por região,
> # usando a função mean (média)
> media <- aggregate(estados$analfabetismo, by=list(estados$reg), FUN=mean)
> # para alterar os nomes das colunas
> names(media) <- c("regiao", "taxa")
 > # Ordenamos o resultado por taxa de analfabetismo
> media <- media[order(media$taxa),]
> View(media) # resulta na tabela 1
> # plotando o gráfico de barras
> barplot(media$taxa, names.arg=media$regiao, col=cor)
> title("Analfabetismo nos EUA / por região"
> # resultado no gráfico abaixo
> # Ordenamos o resultado por taxa de analfabetismo
> media <- media[order(media$taxa),]
> View(media) # resulta na tabela 1
> # plotando o gráfico de barras
> barplot(media$taxa, names.arg=media$regiao, col=cor)
> title("Analfabetismo nos EUA / por região"
> # resultado no gráfico abaixo
O código acima gera o gráfico:

Função pie()
Gráficos de setores ou gráficos de pizza (pie charts) também são úteis para a representação de dados.
pie(x, labels = names(x), edges = 200, clockwise = FALSE,
init.angle = if(clockwise) 90 else 0,
col = NULL, main = NULL, ...)
| x | vetor de valores, exibidos como áreas dos setores no gráfico. |
| labels | nomes para legendas dos setores. NA ou “” = sem legenda. |
| edges | borda externa é um polígono com este número de lados. |
| clockwise | booleano. Sentido horário ou não das fatias. |
| init.angle | ângulo inicial (da primeira fatia). |
| col | vetor de cores de preenchimento das fatias. |
| main | título do gráfico. |
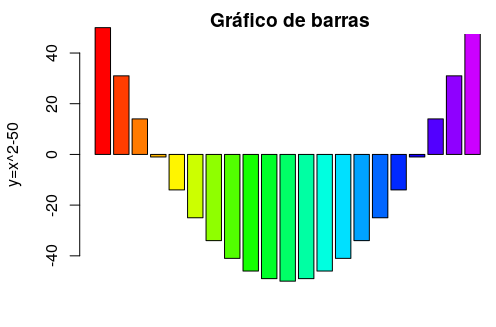
> z <- (-10:10)^2 - 50
> barplot(z, col=rainbow(25), main="Gráfico de barras", ylab="y=x^2-50")
> dev.new()
> legenda <- paste("fatia ",1:16) # gera vetor fatia 1, ..., fatia 16
> cores <- c("#F3E16E", "#6EC6F3", "#6FF36E", "#F36E84")
> pie(rep(1,16), col=cores, labels=legenda, main="Setores")
O seguinte gráfico é gerado:

O código abaixo gera três gráficos de setores (pie charts). As populações listadas para os cinco países mais populosos são dadas em milhões. No gráfico-2 os percentuais (apenas entre estes 5 países) são exibidas. No terceiro gráfico a package plotrix é usada para desenhar um gráfico em 3 dimensões.
> populacao <- c(1420, 1368, 329, 269, 212)
> pais <- c("China", "India", "EUA", "Indonesia" , "Brasil")
> pie(populacao, labels=pais, main = "população em milhões")
> # Gera Gráfico-1
> pc <- round(populacao/sum(populacao)*100)
> pc # porcentagem de população (entre estes 5 países)
[1] 39 38 9 7 6
> label <- paste(pais, "-", pc, "%", sep="")
> label
[1] "China-39%" "India-38%" "EUA-9%" "Indonesia-7%" "Brasil-6%"
> pie(populacao, labels=label, col=rainbow(length(labels)), main = "população em milhões (%)")
> # Observe que length(labels)=5 e temos 5 cores geradas
> # Gera Gráfico-2
> library(plotrix) # deve ser instalado com install.packages("plotrix")
> pie3D(populacao, labels=label,explode=0.1, main="3D Gráfico setores")
> # Gera Gráfico-3

Os gráficos de setores são podem ser úteis para uma visualização rápida de uma relação entre valores. No entanto podem dificultar a análise mais minuciosa destes dados. Por exemplo, se dois setores tem aproximadamente o mesma área pode ser difícil perceber qual é maior. Em geral o uso de barras é mais recomendado.
Uma alternativa atraente é o fan.plot, carregado junto com a library plotrix. Neste tipo de gráfico os setores são sobrepostos e seus raios variados para que todos apareçam na representação.
> library(plotrix) > populacao <- c(1420, 1368, 329, 269, 212) > pais <- c("China", "India", "EUA", "Indonesia" , "Brasil") > fan.plot(populacao, labels=pais, main = "Usando o fan.plot", col=rainbow(5)) > # O gráfico à direita é gerado.
Histogramas com a função hist()
Histogramas (ou distribuições de frequências) são uma forma de exibir a distribuição de uma variável contínua. A faixa de valores a serem analisados é dividida em classes (que podem ser ou não uniformes). A base de cada retângulo na representação é dada pela extensão da classe e a altura pela quantidade de dados (frequências) dentro de cada classe. Histogramas são criados com a função hist(v) onde v é um vetor numérico.
O parâmetro freq=FALSE gera um gráfico baseado em densidades de probabilidade e não em frequências. O parâmetro break informa em quantos classes os dados devem ser divididos. Por default as classes são divididas uniformemente.
> distUniforme <- runif(1000, 0, 10) > # gera 1000 valores distribuídos uniformemente, com média 0 e desvio padrão 1 > hist(distUniforme, col=rainbow(10)) > # plota o histograma dessa distribuição > distNormal <- rnorm(100000, 0, sd=2) > # gera 10^5 valores distribuídos aleatóriamente com distribuição normal, > # com média 0 e desvio padrão 2 > hist(distNormal, col=rainbow(12)) > # plota o histograma dessa distribuição

Para os exemplos que se seguem usaremos o data frame faithful, embutido na instalação do R. Este é um data frame contendo 272 observações, registradas em 2 variáveis numéricas: eruptions, tempo de erupção, e waiting intervalo entre erupções ambas em minutos.
> attach(faithful) > hist(eruptions) > # Gera o primeiro histograma abaixo > hist(faithful$eruptions, seq(1.6, 5.2, 0.2), prob=TRUE, col=rainbow(18)) > lines(density(faithful$eruptions, bw=0.1)) > rug(faithful$eruptions) > # Gera o segundo histograma abaixo > # Os efeitos de lines() e rug() estão marcados no gráfico > detach(faithful)

Gráficos de densidade kernel
Dada uma variável aleatória discreta, a estimativa de densidade kernel (EDK) é uma técnica para se estimar a função de densidade de probabilidade que melhor se ajusta à esta variável. Ela busca suavizar dados discretos fazendo inferências sobre uma amostra finita de dados. Desta forma é possível extrapolar dados discretos, fazendo previsões para valores não diretamente medidos. O kernel (ou núcleo) é uma função simétrica, suave. Tipicamente se usa a gaussiana, embora outras funções podem ser escolhidas. No R um gráfico de densidade kernel para o vetor x pode ser obtido com plot(density(x, )).
> attach(mtcars) > # Construimos uma densidade usando > # mtcars$mpg (milhas por galão) > d <- density(mpg) > plot(d, main="Milhas/galão (densidade kernel)") > # Para colorir a área sob a curva > polygon(d, col="lightblue", border="black") > # Para inserir marcas nos valores > # discretos que geraram a densidade > rug(mpg, col="red") > detach(mtcars)
A função polygon() desenha um polígono com vértices x, y, neste caso os pares fornecidos pela densidade. rug() marca os valores presentes no vetor mtcars$mpg.
Gráficos de densidade kernel podem ser usados para comparar dados em grupos distintos. Para isso usaremos o pacote sm. Nesse pacote usamos
a função sm.density.compare() para sobrepor gráficos nos grupos dentro de fatores cyl.f, que são, no caso, 4, 6 e 8. O formato é sm.density.compare(x, factor) onde x é um vetor numérico e o fator fornece a variável de agrupamento.
> install.package("sm")
> library(sm)
> attach(mtcars)
> cyl.f <- factor(cyl, levels= c(4,6,8),
labels = c("4 cilindros", "6 cilindros", "8 cilindros"))
> sm.density.compare(mpg, cyl, xlab="Milhas por galão")
> title(main="Consumo x Cilindros")
> cores<-c(2:(1+length(levels(cyl.f))))
> legend(locator(1), levels(cyl.f), fill=cores)
> # locator(1) faz com que o quadro de legendas
> # fique ancorado no ponto clicado pelo usuário.
> detach(mtcars)
O código acima gera o gráfico:

Gráficos de caixas boxplot()
Um gráficos de caixas (boxplot()) é uma ferramenta muita usada para analisar e comparar a variação de uma variável entre diferentes grupos de dados. Ela representa uma variável traçando as mesmas informações obtidas em sumário de cinco números:
o mínimo, o quartil inferior (percentil 25), a mediana (percentil 50), o quartil superior (percentil 75) e o máximo. Ele também pode ser usado para mostrar outliers (ou discrepantes, que são valores fora do intervalo de ± 1,5 * IQR, onde IQR é o intervalo interquartil definido como o quartil superior menos o menor quartil).
Aproveitamos, nas linhas de código abaixo, para rever algumas funções estatísticas básicas, como median(), quantil() e summary().
> u <- mtcars$mpg
> min(u)
[1] 10.4
> max(u)
[1] 33.9
> median(u)
[1] 19.2
> quantile(u)
0% 25% 50% 75% 100%
10.400 15.425 19.200 22.800 33.900
> quantile(u, .25)
25%
15.425
> quantile(u, .75)
75%
22.8
> quantile(u, .25, .5,.75)
25%
15.425
> quantile(u, c(.25, .5, .75))
25% 50% 75%
15.425 19.200 22.800
> fivenum(u)
[1] 10.40 15.35 19.20 22.80 33.90
> summary(u)
Min. 1st Qu. Median Mean 3rd Qu. Max.
10.40 15.43 19.20 20.09 22.80 33.90
> boxplot(u, main="Box plot", ylab="Milhas/galão")
O gráfico é gerado:

Boxplots podem ser usados para comparar grupos de variáveis dentro de um dataframe ou lista. O formato para isto é: boxplot(formula, data=dataframe) onde formula é uma relação entre campos do dataframe. Um exemplo de fórmula é y ~ A, onde A é uma variável categórica. Neste caso um plot separado de y é traçado para cada valor de A. A fórmula y ~ A*B resultaria em plots separados de y para cada combinação dos níveis nas variáveis categóricas A e B.
> boxplot(mpg ~ cyl, data=mtcars,
main="Dados de Consumo",
xlab="Número de Cilindros",
ylab="Milhas/galão", col=c("red", "blue", "green"), varwidth=TRUE)
> legend(locator(1), levels(cyl.f), fill=c("red", "blue", "green"))

O opção varwidth=TRUE faz com que as caixas tenham larguras proportionais à raiz quadrada do tamanho das amostras. O parâmetro horizontal=TRUE (não usado no gráfico acima) produz a reversão da orientação dos eixos.
Visualizações interativas
O R fornece muitas formas de exibir gráficos que podem ser modificados por interações com o usuário. Vamos exibir aqui apenas alguns exemplos.
Gráficos interativos com iplots
> library(iplots)
> attach(mtcars)
> cyl.f <- factor(cyl)
> gear.f <- factor(gear)
> ihist(mpg) # histograma
> ibar(carb) # gráfico de barras
> iplot(mpg, wt) # gráfico de pontos
> ibox(mtcars[c("qsec","disp","hp")]) # boxplots
> ipcp(mtcars[c("mpg","wt","hp")]) # coordenadas paralelas
> imosaic(cyl.f,gear.f) # gráfico mosaico

ihist(mpg). O colorido foi feito após a geração do gráfico usando-se o item de menu View > Set color(rainbow).Leaflet
O leaflet é uma biblioteca javascript voltada para a visualização interativa de mapas. O código abaixo carrega uma sessão com o leaflet. A função addTiles() insere uma camada com um mapa ao leaflet inicializado.
> library(dplyr)
> library(leaflet)
> leaflet() %>% addTiles()
> # O gráfico 1 é desenhado.
> # Inserindo a latitude e a longitude da
> # Praça da Liberdade, em Belo Horizonte, MG.
> # (que foi encontrada no Google Maps)
> pcaLiberdade <- data.frame(longitude = -43.938023, latitude= -19.931743)
> pcaLiberdade$titulo <- "Praça da Liberdade, BH!"
> # pcaLiberdade é um data frame com campos:
> pcaLiberdade
longitude latitude titulo
1 -43.93802 -19.93174 Praça da Liberdade, BH!
> leaflet(pcaLiberdade)
%>% addTiles()
%>% addMarkers(lat = ~latitude, lng = ~longitude, popup = ~titulo)
> # O gráfico 2 é desenhado

dplyr e leaflet devem ser instaladas. Observe as linhas de retorno na instalação para verificar sucesso ou erro. A biblioteca iplots, por exemplo, depende de Java.
Biblioteca shiny
> library(shiny)
> ui <- basicPage(
plotOutput("plot1", click = "plot_click"),
verbatimTextOutput("info")
)
> server <- function(input, output) {
output$plot1 <- renderPlot({
plot(mtcars$wt, mtcars$mpg)
})
output$info <- renderText({
paste0("x=", input$plot_click$x, "\ny=", input$plot_click$y)
})
}
> shinyApp(ui, server)
Listening on http://127.0.0.1:6260
A url mostrada (no caso “http://127.0.0.1:6260”) deve ser visualizada no Browser. A cada clique de mouse as coordenadas do cursor são exibidas na caixa abaixo, como mostra a figura.

Biblioteca plotly
O código abaixo utiliza o data frame diamonds que contém informações sobre cor, clareza, medidas, carat, preço de diamantes. O ponto clicado abre um pop-up com dados sobre a posição no gráfico e a clareza do diamante.
> library(plotly)
> set.seed(100)
> d <- diamonds[sample(nrow(diamonds), 1000), ]
> plot_ly(d, x = carat, y = price, text = paste("Clareza: ", clarity),
mode = "markers", color = carat, size = carat)

Sobre operadores em R
Em R o programador pode criar aperadores ou alterar o significado de operadores nativos usando o sinal “`” (backtick ou acento grave).
Por exemplo:
`+` <- function(a, b) paste(a, b, sep="")
"a"+"v" # retorna "av"
O sinal "+" se transformou no operador de concatenação.
Em geral se pode programar %X% (qualquer X) para qualquer funcionalidade.
`%@%` <- function(a, b) a^b
`%*%` <- function(x, y) x/y
2 %@% 3 # retorna 8
15 %*% 3 # retorna 5
As bibliotecas magrittr e dplyr definem o operador %>% com o seguinte significado:
`%>%` <- function(x, FUN) FUN(x)
Isso quer dizer que
x %>% hist
é o mesmo que
hist(x)
Por exemplo:
iris$Sepal.Length %>% hist # traça o histograma do vetor
mtcars$mpg %>% hist(col=rainbow(5)) # histograma de mtcars$mpg usando 5 cores.
Gráficos tridimensionais
Existem muitas bibliotecas em R para a geração de gráficos 3D. Entre eles estão: RGL, car, lattice e scatterplot3d (e muitos outras).
Gráfico de dispersão em 3D com scatterplot3d
scatterplot3d é uma biblioteca de uso simples, com formato básico:
scatterplot3d(x, y=NULL, z=NULL)
onde x, y, z são as coordenadas dos pontos a serem plotados. Os argumentos y e z são opcionais, dependendo da estrutura de x.
- Se x é uma fórmula (como em
zvar~xvar+yvar) entãoxvar,yvarezvarsão usados como valores para x, y e z. - Se x é uma matriz com pelo menos 3 colunas então as variáveis x, y e z são lidas diretamente da matriz.
> library("scatterplot3d")
> data(iris)
> flor <- iris[1:50,1:3] # 50 linhas, 3 primeiras colunas
> names(flor) <- c("comprimentoSepala", "larguraSepala", "comprimentoPepala")
> # A forma mais simples de uso:
> scatterplot3d(flor)
> scatterplot3d(flor, pch = 20,
main="Gráfico dispersão 3D",
xlab = "Comprimento sétala (cm)",
ylab = "Largura sétala (cm)",
zlab = "Comprimento pétala (cm)", color="steelblue")
> # O gráfico 1 é gerado. (pch=20 usa símbolo bola cheia)
> scatterplot3d(flor, pch = 8, main="pch = 8 -> estrela",
color="#E8582D", angle=55, grid=TRUE, box=FALSE)
> # O gráfico 2 é gerado. pch=8 usa símbolo estrela,
> # o gráfico é girado de 55º, com grid e sem a caixa envelope.

> z <- seq(-10, 10, 0.01)
> x <- cos(z)
> y <- sin(z)
> scatterplot3d(x, y, z, highlight.3d = T, col.axis = "blue",
col.grid = "lightblue", main = "Hélice", pch = 20)

O gráfico ao lado é gerado.
Mais informações sobre scatterplot3d no site STHDA.
Mais informações sobre 3d scatterplots no site STHDA.
Outras visualizações em 3D com scatter3d
O formato básico para scatter3d, com alguns de seus parâmetros, é o seguinte:
scatter3d(formula, data, subset, radius, xlab, ylab, zlab, ...)
ou
scatter3d(x, y, z,
xlab, ylab, zlab, revolutions=0, speed=1,
bg.col=c("white", "black"), axis.scales=TRUE,
axis.col, surface=TRUE, surface.col=carPalette()[-1],
fill=TRUE, point.col="yellow", text.col=axis.col,
radius=1, groups=NULL, fill=TRUE, grid=TRUE,
ellipsoid=FALSE, sphere.size=1, radius=1, threshold=0.01,
parallel=TRUE, ellipsoid=FALSE, id=FALSE, ...)
onde
| formula | fórmula y ~ x + z. Para plotar os pontos por grupos use y ~ x + z | g onde g é o fator que distingue grupos. |
| data | data frame usado para avaliação da fórmula. |
| x, y, z | coordenadas dos pontos a serem plotados. Os argumentos y e z são opcionais, dependendo da estrutura de x. |
| subset | expressão definindo subconjunto das observações a serem usadas. |
| xlab, ylab, zlab | labels nos eixos. |
| radius | raios das esferas representando pontos. |
| axis.scales | Se TRUE, nomeia valores nas pontas dos eixos. |
| revolutions | quantas revoluções a figura fará (animação). |
| bg.col | cor de fundo. |
| axis.col | cores para eixos. |
| surface.col | vetor de cores para os planos. |
| point.col | cores dos pontos. |
| text.col | cores dos eixos. |
| grid.col | colour of grid lines on the regression surface(s). |
| surface | lógico, plotar superfícies. |
| fill | lógico. Preencher superfícies com cores. |
| grid | lógico. Plotar lines da grade nas superfícies de regressão. |
| grid.lines | número de linhas nas grades. |
| speed | velocidade de revolução. |
| fov | controla ângulo da perspectiva. |
| groups | Se NULL nenhum grupo é definido. Se um fator uma superfície diferente é desenhada para cada nível. |
| parallel | lógico. Se as superfícies para grupos devem ser paralelas. |
| ellipsoid | lógico. Concentração elipsóide para pontos. |
| labels | texto para labels nos pontos. Default são os índices da observação. |
| col | cores para labels em pontos. |
| … | outros argumentos. |
> install.packages(c("rgl", "car"))
> library(rgl, car)
> data(iris)
> sep.l <- iris$Sepal.Length
> sep.w <- iris$Sepal.Width
> pet.l <- iris$Petal.Length
> scatter3d(x = sep.l, y = pet.l, z = sep.w) # plota Gráfico-1
> scatter3d(x = sep.l, y = pet.l, z = sep.w,
point.col = "steelblue", surface=FALSE) # plota Gráfico-2
> scatter3d(x = sep.l, y = pet.l, z = sep.w,
groups = iris$Species) # plota Gráfico-3
> scatter3d(x = sep.l, y = pet.l, z = sep.w, groups = iris$Species,
grid = FALSE, fit = "smooth") # plota Gráfico-4
> scatter3d(x = sep.l, y = pet.l, z = sep.w, groups = iris$Species,
surface=FALSE, ellipsoid = TRUE) # plota Gráfico-5
> scatter3d(x = sep.l, y = pet.l, z = sep.w,
groups = iris$Species, surface=FALSE,
grid = FALSE, ellipsoid = TRUE) # plota Gráfico-6
> # Para gravar estes gráficos nos formatos png e pdf podemos usar
> rgl.snapshot(filename = "plot.png")
> rgl.postscript("plot.pdf",fmt="pdf")
Todos os gráficos podem ser girados e redimensionados com o arraste de mouse.


Continuaremos o estudo sobre gráficos na próxima sessão, usando ggplot2.
Thank you for the auspicious writeup. It in fact was a amusement account it.
Look advanced to more added agreeable from you!
However, how can we communicate?